

施一公研究组发文报道酵母B*剪接体电镜结构 捕获最后一个剪接体基本状态
| 来源:清华新闻网【字号:大 中 小】
清华新闻网3月15日电 3月15日,清华大学生命学院施一公教授研究组就剪接体的机理与结构研究,于《细胞》(Cell)期刊发表题为《催化激活状态的酵母剪接体结构揭示RNA剪接分支反应的机理》(Structures of the Catalytically Activated Yeast Spliceosome Reveal the Mechanism of Branching)的科研论文,揭示了剪接体第一步剪接反应前的瞬变状态——催化激活剪接体(catalytically activated spliceosome,定义为“B*复合物”)4个不同构象的高分辨率三维结构,这是目前RNA剪接循环中最后一个未被解析的基本状态。至此,施一公研究组成为世界上首个、也是唯一一个成功捕获并解析了RNA剪接过程中所有完全组装剪接体高分辨率三维结构系列成果的团队。该文报道的这4个同一状态却不同构象的剪接体结构,整体分辨率为2.9埃-3.8埃,核心区域的分辨率高达2.7埃,是目前报道的最高分辨率剪接体结构,该结构首次揭示了第一步剪接反应发生过程中的动态变化,展现了剪接因子对于剪接反应发生的重要作用,第一次从结构信息中回答了剪接体对不同pre-mRNA底物识别的特异性等重要科学问题。
1977年,科学家们首次发现来自于腺病毒的mRNA与其对应的DNA转录模板并不能形成连续的杂交双链,而是在杂交双链的不同位置伸出了环状的DNA单链。这个重大发现表明,遗传信息从DNA传递到mRNA上并不只是通过转录,还需要pre-mRNA剪接来进一步完成“无效”遗传信息的去除与有效遗传信息的拼接。“无效”的遗传信息不具有翻译功能,被称为内含子,而可以被核糖体翻译的有效遗传信息叫做外显子,内含子被去除、外显子被连接这一过程即为RNA剪接。RNA剪接普遍存在于真核生物中,随着物种的进化,含有内含子的基因数量增加,发生RNA剪接的频率也相应增高,使得一个基因编码多个蛋白质成为可能,极大的丰富了蛋白质组的多样性,也是真核生物多样性的重要原因之一。
RNA剪接是真核生物基因表达调控的重要环节之一,其化学本质是两步转酯反应。这种看似简单的化学反应在细胞中难以自行发生,而负责执行这一化学反应的是细胞核内一个巨大且高度动态变化的分子机器——剪接体(spliceosome)。从1977年首次发现RNA剪接至本世纪初,科学家们通过免疫沉淀、基因敲除、交联质谱、建立体外剪接反应系统等研究手段,初步建立起剪接体的组装、激活与解聚的发生过程,以及蛋白与蛋白、蛋白与核酸之间的相互作用、相互调控等复杂的RNA剪接调控网络。在剪接反应过程中,多种蛋白-核酸复合物及剪接因子按照高度精确的顺序发生结合、重排和解聚,依次形成预组装复合物U4/U6.U5 Tri-snRNP(U4/U6.U5三小核核糖核蛋白复合物)以及至少8个状态的完全组装剪接体pre-B、B、Bact、B*、C、C*、P以及ILS复合物(图1)。

图1 RNA剪接示意图
(图片来源: Yan, C., Wan, R., & Shi, Y. (2019).Cold Spring Harbor perspectives in biology, 11(1), a032409.)
由于剪接体高度的动态性和复杂性,获得不同状态的剪接体的高分辨率三维结构被公认为世界难题。在这种巨大的挑战下,施一公教授率领研究组迎难而上,经过7年的努力,终于在2015年首次报道了裂殖酵母剪接体3.6埃的高分辨率结构,首次展示了剪接体催化中心近原子分辨率的结构。这一重大研究成果对RNA剪接机理的研究产生革命性影响。自2015年第一个剪接体结构发表以后,施一公研究组相继解析了酿酒酵母剪接体复合物处于8个不同状态的高分辨率结构,分别是3.8埃的预组装复合物U4/U6.U5 Tri-snRNP、3.3埃-4.6埃的预催化剪接体前体pre-B complex、3.9埃的预催化剪接体B complex、3.5埃的激活状态剪接体Bact complex、3.4埃的第一步催化反应后剪接体C complex、4.0埃的第二步催化激活剪接体C* complex、3.6埃的完成两步转酯反应后的剪接体P complex,以及3.5埃的内含子套索剪接体ILS complex的结构。这些已解析的剪接体基本覆盖了整个RNA剪接循环,从分子层面揭示了剪接体催化RNA剪接两步反应的工作机理,同时为理解剪接体的组装、激活和解聚等过程的发生提供结构依据。然而,最后一个未被解析的完全组装剪接体B* complex,对于理解第一步剪接反应的发生具有至关重要的作用。由于其高度的动态性与瞬时性,如何捕获B* complex成为领域内的一大难题,也是世界上不同课题组争先解决的问题之一,捕获并解析其结构迫在眉睫。
尽管之前积累了大量剪接体结构研究的经验,施一公研究组在针对如何捕获稳定的B* complex时,却是举步维艰,困难重重。由于B* complex和C complex是第一步剪接反应发生时一前一后的两个状态,领域内对其鉴定的核心区别在于5’剪接位点与分支点之间的磷酸二脂键是否形成。因此,在不影响剪接体正常组装和激活的前提下,如何阻止第一步反应发生,并且保持5’剪接位点与分支点已经进入RNA剪接的活性反应中心,并形成反应前的活性位点,成为本文研究的最大难点。施一公研究组分别进行了体内内源直接阻断和体外建立剪接反应阻断等方法的尝试,最终都无法获得稳定的B* complex样品。在最新发表的这篇《细胞》文章中,施一公研究组将体内阻断与体外建立剪接反应相结合,对提纯方案多次探索,最终优化出一套可以获得稳定的、性质良好的B* complex样品。为了探索剪接体对不同pre-mRNA底物的识别特异性,施一公研究组选取了领域内常用的两种pre-mRNA作为剪接体结合的底物,随后利用单颗粒冷冻电镜技术重构出了4个不同构象B* complex整体分辨率为2.9埃-3.8埃的冷冻电镜结构,并搭建了原子模型,其中包含了4条RNA和35个蛋白(图2)。
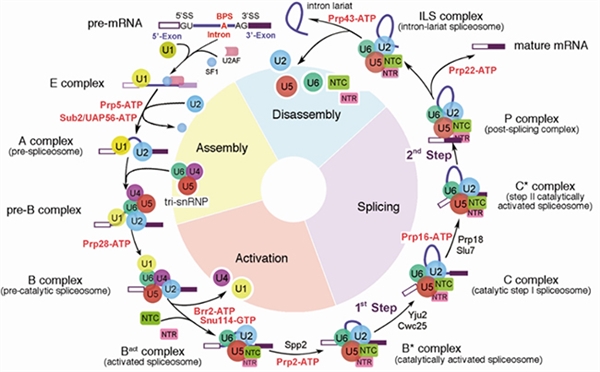
图2 酿酒酵母催化激活剪接体4个构象的三维结构
该文解析的4个不同构象的B* complex结构,首次展示了RNA剪接第一步反应发生过程中分支点如何被剪接体逐步“推入”活性反应中心的动态变化,其中第一步剪接因子Yju2在稳定分支点中发挥了举足轻重的作用,尽管此时5’剪接位点与分支点的距离十分接近(4.3埃),却不足以形成磷酸二脂键。从而,该结构也清晰地揭示了关键蛋白因子Cwc25在激发第一步剪接反应中的重要作用(图3)。值得一提的是,在这个结构中,第一次观察到了pre-mRNA中分支点A被5’剪接位点GU通过经典的碱基互补配对以及碱基堆积力共同识别的机制,这个结构信息进一步证明了该位点在真核生物中高度保守的重要性。本文另一大亮点是首次揭示了剪接体对不同pre-mRNA底物识别的特异性,为体内不同pre-mRNA的剪接效率存在差异提供了最直接的结构信息。
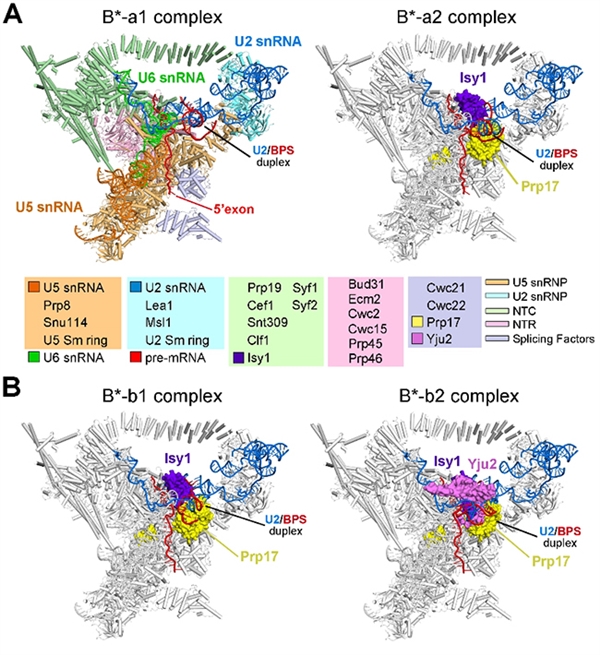
图3 酿酒酵母剪接体介导分支反应的模型
截至目前为止,施一公研究组在酵母中一共解析了10个不同状态的剪接体高分辨的三维结构(如图4),成果全部发表于国际顶级期刊《科学》和《细胞》(Science 7篇,Cell 3篇),从组装到被激活,从发生两步转酯反应发生到剪接体的解聚,这10个状态的剪接体完整覆盖了剪接通路,首次将剪接体介导的RNA剪接过程完整的串联起来,为理解RNA剪接的分子机理提供了最清晰、最全面的结构信息。(观看完整视频请点文末链接)
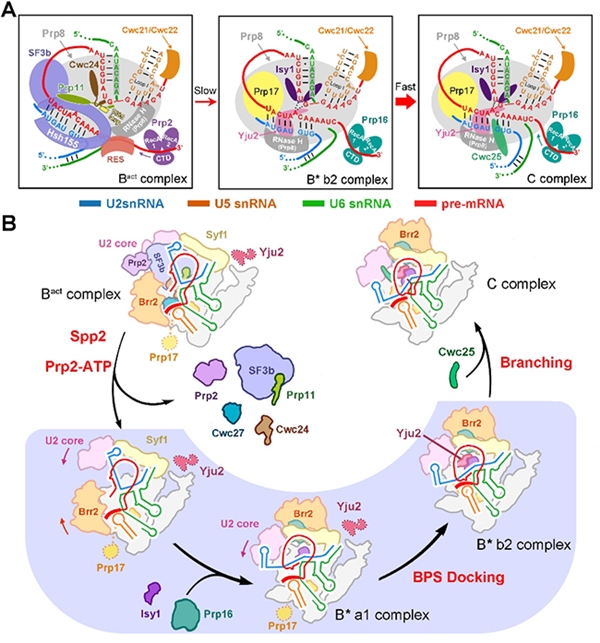
图4 施一公研究组解析的酵母剪接体结构汇总
(图片来源: https://ygshi.org/research)
清华大学生命学院施一公教授为本文的通讯作者;清华大学医学院博士后、高精尖创新中心卓越学者万蕊雪、生命学院四年级博士研究生白蕊为该文的共同第一作者,清华大学生命学院博士后、高精尖创新中心卓越学者闫创业为结构模型的搭建提供了帮助;清华大学冷冻电镜平台主管雷建林博士为冷冻电镜数据收集提供了帮助。电镜数据采集于清华大学冷冻电镜平台,计算工作得到清华大学高性能计算平台、国家蛋白质设施实验技术中心(北京)的支持。本工作获得了北京结构生物学高精尖创新中心(清华)及国家自然科学基金委的经费支持。